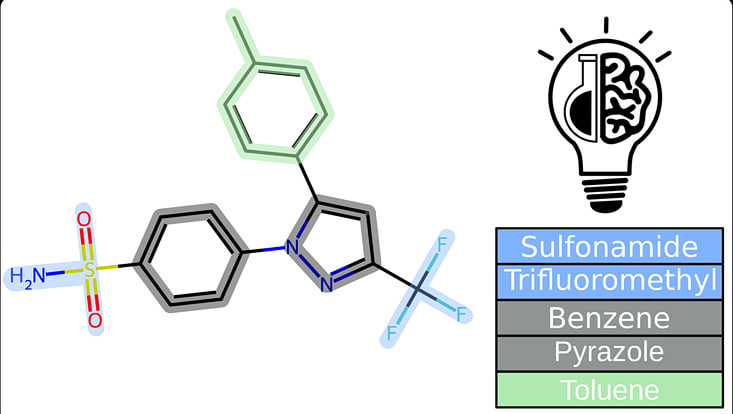
Algorithmisches Molekulares Design

Arbeitsgruppenleiter: Prof. Dr. M. Rarey
Die Entwicklung neuer bioaktiver Moleküle, wie sie beispielsweise als Wirkstoffe in Medikamenten vorkommen, findet im zunehmenden Maße mit dem Computer statt. Die Entwicklung der dazu notwendigen Softwaresysteme ist der Forschungsschwerpunkt der Arbeitsgruppe AMD am Zentrum für Bioinformatik. Die dabei auftretenden Fragestellungen sind vielfältig und deren Bearbeitung verlangt oftmals nach ausgefeilten, maßgeschneiderten Methoden. Zur Lösung bioinformatischer Fragestellungen setzen wir ein großes Spektrum an Informatik-Techniken, vom Algorithmenentwurf über Machine Learning, von modernen Software-Engineering und User-Interface Design zu performanten Datenbanktechniken und Webtechnologien ein. Wir haben den Anspruch, Softwaresysteme mit hoher praktischer Relevanz zu konstruieren und für Naturwissenschaftler zur Verfügung zu stellen .