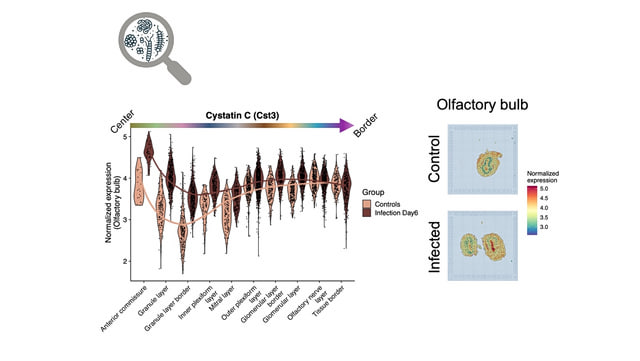
Foto: Kern et al. “Spatiotemporal transcriptomic niches of complement pathway and serine protease inhibitor activation in aging and infection.” In: bioRxiv (2024), doi: 10.1101/2024.11.04.621811. Lizenziert unter CC BY-NC-ND 4.0.
Unsere Arbeitsgruppe engagiert sich in der molekularbiologischen Grundlagenforschung mit dem Ziel, zelluläre und regulatorische Prozesse datengetrieben zu verstehen. Wir analysieren, oft in nationaler und internationaler Kooperation, große molekulare Datensätze – etwa aus RNA-Sequenzierung (Bulk und Single-cell) – und untersuchen grundlegende Fragestellungen zur Genregulation, Zelltypidentität, molekularen Netzwerken und Krankheitsmechanismen in Gesundheit, Alterung und Krankheit.
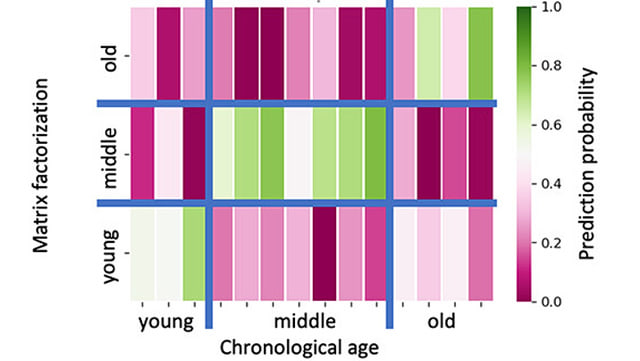
Beispiel: Hochdurchsatz single-cell und spatial Transkriptomik
Die Arbeitsgruppe beschäftigt sich mit der Analyse hochdimensionaler molekularer Daten, die durch moderne High-Throughput-Verfahren generiert werden – sowohl in Bulk- als auch in Single-Cell-Experimenten. Dabei untersuchen wir insbesondere Gen- und miRNA-Expressionsprofile, um biologische Prozesse auf transkriptioneller Ebene besser zu verstehen.
Bulk-RNA-Sequenzierungen liefern robuste Mittelwerte über Zellpopulationen und sind ein etabliertes Werkzeug zur Analyse differenziell exprimierter Gene und regulatorischer miRNAs in verschiedenen biologischen Zuständen. Ergänzend dazu ermöglichen Single-Cell- und Single-Nucleus-RNA-Sequenzierungen die Analyse individueller Zellen und eröffnen Einblicke in zelluläre Heterogenität, seltene Zelltypen und dynamische Genexpressionsmuster auf Einzelzellebene. Letztlich haben wir auch bereits Erfahrungen gesammelt im recht jungen Forschungsfeld der Spatial-Omics Technologien, die es erlauben räumliche und zeitliche Koordinaten mit molekularen Messwerten zu verknüpfen.
Beispiel: Neurodegeneration und Infektionsforschung
Wir beschäftigen uns ebenfalls mit der bioinformatischen Analyse molekularbiologischer Daten im Schnittstellenbereich neurodegenerativer Erkrankungen und Infektionskrankheiten. Im Zentrum stehen dabei Krankheiten wie Alzheimer, Parkinson, sowie virale Infektionen, etwa durch SARS-CoV-2. Ziel ist es, die zugrunde liegenden molekularen Mechanismen besser zu verstehen und krankheitsassoziierte Veränderungen auf Zell- und Gewebeebene systematisch zu erfassen.
Für alle diese Datentypen und -modalitäten nutzen wir klassische bioinformatische Werkzeuge – z. B. zur Qualitätskontrolle, Normalisierung und statistischen Analyse – sowie moderne Methoden des Maschinellen Lernens und Deep Learnings. Diese helfen uns, komplexe Muster zu erkennen, Zelltypen zu identifizieren, genregulatorische Netzwerke zu rekonstruieren und krankheitsspezifische Signaturen sichtbar zu machen. Derartige Signaturen erlauben es uns Krankheitsprozesse an ihrer Wurzel zu beschreiben und das translationale Potential für die Entwicklung von neuen Biomarkern oder sogenannter Targets für neue Wirkstoffe systematisch abzuleiten. Durch die Kombination klassischer Analyseverfahren mit modernen datengetriebenen Ansätzen leisten wir einen Beitrag zum besseren Verständnis alters- und krankheitsbedingter Veränderungen im menschlichen Gehirn sowie zur Erforschung immunologischer Prozesse bei Infektionen.